然而,小分子與 RNA 的結合通常親和力不高、結合時間短暫,且在細胞內複雜環境中更難被直接偵測。因此,如何在「活細胞」中追蹤、定位、並定量小分子與 RNA 的結合位置,一直是 RNA 藥物研究的重要挑戰。
為了解決這個問題,Tong 等人(2024)發展並優化了一套完整流程,能夠在**全轉錄體範圍(transcriptome-wide)**探測小分子 RNA 的結合位點 — — 即 Chemical Cross-Linking and Isolation by Pull-down(Chem-CLIP) 與其擴增平台。
本篇文章將介紹此方法的核心概念、實驗流程與其對 RNA 靶向藥物研究的推進。
一、Chem-CLIP:捕捉小分子與 RNA 的瞬時互動
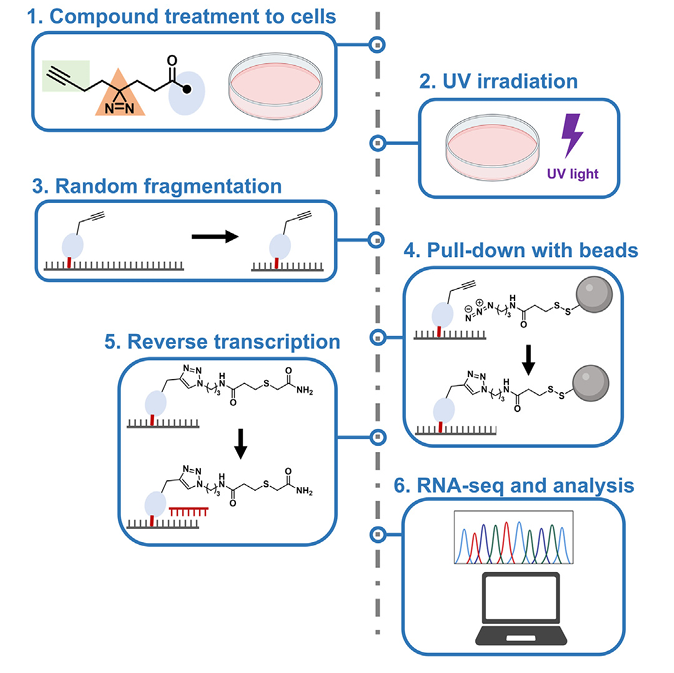
Chem-CLIP 的核心概念非常優雅: 將小分子修飾為三合一探針(probe):
- RNA-binding module:負責與目標 RNA 結合
- diazirine 光交聯基團:經 UV 照射後產生自由基,與鄰近 RNA 共價鍵結
- alkyne 手把(handle):作為後續「click 反應」的標籤,用於拉取(pull-down)
當探針進入細胞並結合 RNA 後,經 UV(350–365 nm)照射便會產生交聯,將原本短暫、可逆的小分子–RNA 結合永久固定,使其得以被分離、富集並進行後續分析。
二、全轉錄體結合位點的偵測策略
論文提出三大應用模式:
1. In vitro 結合占據率(target occupancy)量測
- 使用放射性標記的 RNA
- 與 Chem-CLIP probe 混合、照光
- pull-down 後定量 RNA 捕捉比例 → 可用於比較不同化合物的結合能力或測試特異性。
2. 在活細胞中建立 RNA「可被小分子結合」的圖譜(ligandability map)
步驟如下:
- 細胞處理小分子 Chem-CLIP probe
- UV 交聯
- 提取總 RNA
- 隨機打斷 RNA(約 150 nt)
- 透過 alkyne–azide click chemistry 富集被交聯的 RNA
- RNA-seq 分析
結果能反映:
- 哪些 RNA(例如特定非編碼 RNA、重複序列、特定轉錄本)傾向與小分子互動
- 結合的相對強度
3. 小分子結合位點的精確定位(binding-site mapping)
由於小分子與 RNA 的交聯會阻礙逆轉錄(RT),造成 cDNA 的截斷(RT stop),因此序列讀段起始位置便能反映小分子在 RNA 上的結合位點。
這讓研究者能在全轉錄體尺度下解析:
- 小分子偏好與 RNA 哪種結構互動(例如 stem-loop、G-quadruplex)
- 結合位點是否位於病理序列,例如重複擴增序列(CUG、CAG repeats)
三、技術優勢與創新之處
1. 捕捉細胞內真實結合事件
傳統 RNA-binding assays 多採 in vitro 條件,無法反映細胞內 RNA 的折疊與動態。 Chem-CLIP 在「真實的細胞環境」中記錄小分子–RNA 的天然互動。
2. 能處理親和力低、結合短暫的小分子
光交聯將短暫結合固定化,使研究者可檢測到小分子片段(fragment-like molecules)也可能的弱互動。
3. 全轉錄體規模、可定址的精確結構資訊
不僅能知道小分子與哪些 RNA 結合,還能知道其結合於 RNA 的哪個結構區域。
4. 適用於小分子藥物發現
- 篩選 fragment libraries
- 表徵 lead compounds 的細胞內靶點
- 避免 off-target RNA 交互作用
四、方法的限制與注意事項
- UV 交聯效率受 RNA 結構與距離影響,可能產生偏倚
- Diazirine 有時會與非 RNA 標的交聯
- 需有適當的對照組(probe without RNA-binding module)
- 低豐度 RNA 的偵測敏感度可能有限
- 高度重複序列的 mapping 較具挑戰
五、研究前景與應用方向
Chem-CLIP 及其衍生技術為 RNA 藥物研究開創全新可能:
- 系統性 mapping RNA 上可被小分子攻擊的「熱點(hotspots)」
- 揭示 RNA 結構生物學的新地景
- 開發治療 RNA 重複擴增疾病(如 DM1、亨丁頓舞蹈症)的新策略
- 精準解析 off-target 作用以提升藥物安全性
在 RNA 成為下一代藥物標靶的趨勢下,此技術將具有高度戰略價值。